[文献精读]比较母体血浆和血清中的游离DNA:使用基因组和表观基因组方法来比较数量、质量和组织来源
2018-01-06 14:28来源:原版作者:Clara Pérez-Barrios等
原文出处
Transl Lung Cancer Res 2016;5(6):665-672
作者
Clara Pérez-Barrios1*, Irene Nieto-Alcolado2*, María Torrente3, Carolina Jiménez-Sánchez2, Virginia Calvo3, Lourdes Gutierrez-Sanz3, Magda Palka3, Encarnación Donoso-Navarro1, Mariano Provencio3, Atocha Romero2,3
摘要
背景:在癌症患者中实施用于生物标志物检测和对治疗监测的反应的液体活检可能增加实验室的通过量,需要开发用于循环游离DNA(cfDNA)分离的自动化方法。
方法:本研究比较了MagNA Pure Compact(MPC)核酸分离试剂盒I和Maxwell®RSC(MR)ccfDNA等离子体试剂盒,后来与QIAamp循环核酸(QCNA)试剂盒比较,使用57例来自癌症患者的血浆样品。使用Qubit荧光计测量cfDNA浓度。使用Agilent 2100生物分析仪评估DNA片段长度。循环肿瘤DNA(ctDNA)通过数字PCR(dPCR)定量。
结果:首先,买球网观察到MPC方法比MR显着提取较少的cfDNA(P <0.0001)。然而,QCNA和MR试剂盒的提取率没有显着差异。 cfDNA分离产量也与肿瘤分期相关,但与肿瘤位置无关。其次,在88%的样品中观察到寡核苷酸DNA梯形图,在MPC和MR方法之间观察到单,二和三核小体DNA片段的回收率的显着差异。最后,使用数字PCR对38个配对样品进行了对cfDNA的肿瘤突变定量。配对样本之间的突变等位基因级分(MAF)没有显着差异。
结论:分离cfDNA的方法可以影响DNA产量和分子量分数的恢复。对于常规临床实践中的cfDNA分析,应考虑这些观察结果。
关键字:癌症; 循环游离DNA(cfDNA);表皮生长因子受体(EGFR); KRAS
前言
全球癌症是发病和死亡的主要原因之一。多年来,癌症的发病率有所增加,这一趋势预计在不久的将来会上升(1-3)。理解癌症发展的生物学过程的最新突破已经导致更有效的治疗策略和靶向治疗不可逆转地改变了癌症患者的治疗。然而,使用这种疗法需要在肿瘤活检中进行生物标志物测试,并获得足够量的肿瘤材料用于分析可靶向基因中的体细胞突变是有些挑战的。
由肿瘤细胞释放的循环游离DNA(cfDNA)保留了原始组织的特征。因此,其研究允许通过非侵入性程序对肿瘤的遗传表征,并解决常规活检的困难(4-6)。此外,与固体活组织检查不同,在液体活检中,循环肿瘤DNA(ctDNA)可以在治疗过程中进行定量,这可能有助于测量肿瘤对治疗的反应(7,8)。在这种情况下,要测试的样本数量有望大幅增加,因为开发高通量技术可以使cfDNA分离成为重要的挑战。以这种方式,自动化系统的出现可以提高过程的重复性和鲁棒性,并有助于在临床环境中实施液体活检。
本研究的目的是比较两个自动系统的cfDNA分离的性能,即MagNA Pure Compact(MPC)核酸分离试剂盒I(Roche Diagnostics,Penzberg,Germany)和Maxwell®RSC(MR)ccfDNA等离子体试剂盒(Promega Corporation,Madison,WI,USA),后者与QIAamp循环核酸)Kit(QIAgen,Valencia,CA,USA)手动提取试剂盒,这是最广泛的用于cfDNA分离的方法之一。具体来说,买球网比较了获得的核酸的cfDNA提取产率和片段大小。最后,买球网通过比较来自具有驱动突变的肿瘤患者的38个cfDNA样品中的突变等位基因分数(MAF)测量通过数字PCR(dPCR)来检查肿瘤特异性突变的鉴定是否可受cfDNA分离方法的影响。
方法
样本人群
癌症患者共获得57份样本,并在签署了适当的知情同意书后于2015年2月至6月期间进行了研究。该研究由医院普埃尔塔德耶罗伦理委员会批准。该队列由成年男性和女性诊断患有肺癌或结肠癌,临床III-IV期(表S1)。从临床和病理报告中获得有关人口统计学,临床病理特征和肿瘤突变状态的信息。
实验方法
买球网将57个周边全血样品收集在含有凝胶屏障的4mL PPT TM管(Becton Dickinson)中,以离心分离血浆。所有样品在抽血时间后2小时内在室温下处理。在4℃下以1,500g离心10分钟,将细胞级分与血浆分离。离心后,将55个血浆样品分成两等份1mL,将两个血浆样品分成3份等份1mL(图S1)。将等分试样在-80℃下立即储存直到cfDNA提取。弃去溶血样品进行进一步分析。在进行cfDNA提取的同时,样品在4℃的冰箱中解冻。解冻后,以5,000g进行新的离心20分钟,以确保除去上清液中的杂质。
对于cfDNA分离方法比较,通过MR和QCNA处理31个样品,通过MPC和MR处理24个样品,并将两个血浆样品等分成三个并通过所有三种方法进行处理(图S1)。使用MR仪器(Promega),使用制造商规定的MR ccfDNA等离子体试剂盒分离cfDNA,并根据制造商的说明书使用QCNA试剂盒(QIAgen),并遵循“1mL无细胞DNA定制”程序(除了当蛋白酶K孵育过夜),并在MPC仪器(Roche Diagnostics)上使用MagNA Pure Compact Nucleic Acid Isolation Kit I。
在所有情况下,使用起始体积1mL的血浆提取cfDNA,并在50μL所提供的洗脱缓冲液中洗脱。根据制造商的说明书,使用Qubit dsDNA HS Assay试剂盒(Invitrogen,Life Technologies,CA,USA)在Qubit 2.0荧光计(Invitrogen,Life Technologies,CA,USA)上测量cfDNA浓度。
基于制造商推荐的方案,使用Agilent 2100生物分析仪和高灵敏度DNA芯片(Agilent Technologies Inc.,Palo Alto,CA,USA)进行微流控电泳,以评估50至7,000个碱基对(bp)之间的DNA片段长度。通过dPCR分析cfDNA样品,使用EGFR p.T790M(AHRSROS),p.L858R(AHRSRSV)和p.E746_A750delELREA(AHLJ0XO)中以下突变的罕见突变测定法进行分析。和突变在Quantas 3D数字PCR系统(Applied Biosystems,South San Francisco,CA,USA)的KRAS中的p.G12V(AHX1IHY),p.G12D(AH6R5PI)和p.G13D(AHD2BW0)。对于dPCR,将8μL模板cfDNA与20μL反应体积中的0.5μL上述40×TaqMan测定和10μL的2×QuantStudio 3D Master Mix混合。随后,将15μL加载到QuantStudio 3D Digital PCR 20K芯片中。循环条件如下:96℃初始变性10分钟,56℃2分钟40个循环,98℃30秒,72℃10分钟,最终样品维持在22℃至少30分钟。芯片荧光读取两次。使用QuantStudio®3D Analysis Suite™云软件分析结果。需要时,手动调整每个数据集群的自动呼叫分配。测定结果报告为突变型DNA分子相对于突变体和野生型(wt)DNA分子总和的比例。每次运行都包含了wt对照DNA。
统计分析
定性变量通过其频率分布和定量变量的平均值和标准差(SD)或中位数和四分位数范围进行归纳总结。使用不同方法使用相同血浆样品的不同等分试样的cfDNA浓度产率的非参数比较使用配对数据的Wilcoxon有符号秩检验进行评估。使用Mann-Whitney U检验进行不同临床情况(例如肿瘤来源(肺癌对结肠直肠癌)或肿瘤分期(IV期与其他)的不同临床情况的cfDNA浓度产率的非参数比较。分类变量之间的关联通过McNemar's Test测试。空假设被小于0.05的I型误差拒绝。
循环游离核小体结合DNA片段的相对定量表达为特定片段的浓度与相应样品中cfDNA的总浓度之间的比例。使用不同方法在配对样品中获得的循环游离核小体结合的DNA片段比例之间的比较通过Wilcoxon signrank测试来评估配对数据。对于这一分析,根据多次比较的Bonferroni校正,将显着性水平调整为0.0083。 统计分析采用STATA 9.0 SE进行。
结果
根据方法提取的cfDNA产量的比
研究中包括的人群主要是白种人(95%),年龄从41岁到82岁。在收集的57个血浆样本中,47个样品对应于肺癌患者,10个样品对应于结肠直肠癌。总共测量了从57个血浆样品获得的总共116个样品中的cfDNA浓度。总体而言,定量结果显示癌症患者血浆中的cfDNA浓度范围在0.2至24ng /μL之间。
为了比较,通过MR和QCNA从33个血浆样品中提取cfDNA。该子集样品来自肺癌患者,临床III-IV期,除了一个与诊断为结肠直肠癌的患者相对应的样品外。根据买球网的结果,通过MR方法提取的样品的中值浓度为1.25 ng /μL,QCNA方法提取的样品为1.08 ng /μL,方法之间的产量提取没有显着差异(P =
0.775)。有趣的是,买球网观察到,与非转移患者相比,从IV期癌症患者获得的cfDNA的量显着更高。对于使用MR和QCNA方法分离的cfDNA样品,这是正确的(P = 0.045和P = 0.0173)。关于MR和MPC方法的比较,使用两种方法处理了26个血浆样品。这些血浆样品来自IV期结直肠癌患者和转移性疾病的肺癌患者,除了一名IIIA期患者。有趣的是,买球网的数据显示,MPC提取的显着少于MR(1.154 vs. 2.31; P
<0.0001)。买球网还调查肿瘤位置(肺或结肠)是否可以影响cfDNA分离产率。买球网的数据表明,通过MPC或MR方法处理的样品中,根据肿瘤定位,cfDNA产量没有显着性差异(P = 0.9355和0.4834)。

根据方法比较循环游离核小体结合DNA片段分布
以生物分析仪2100的方式,对从MR和QCNA提取的22个血浆样品中获得的44个cfDNA样品和通过MR和MPC方法提取的17个血浆样品获得的34个cfDNA样品进行了分析。
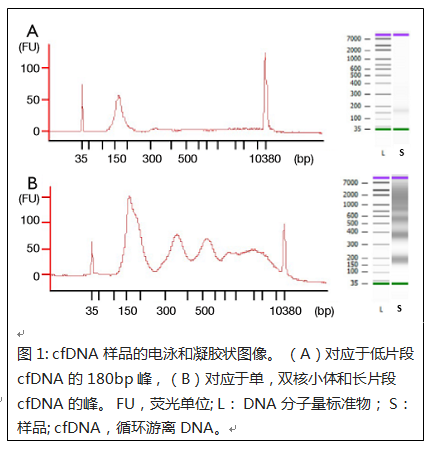
观察到的cfDNA片段的大小为约180bp(平均182bp,范围为151-205bp)。在78个cfDNA样品中的69个中,买球网还观察到寡核苷酸DNA梯形图,因为在电泳测定(图1)中清楚地观察到对应于单,二和三核小体的峰的存在,表明凋亡细胞死亡。买球网还检测到78个cfDNA样品中的54个中的高分子量(> 10,000bp)DNA片段,其可以源于通过坏死死亡的细胞。
此外,寡核苷酸片段的比例根据提取方法而变化(表1)。值得注意的是,通过MPC方法提取的大小为150〜200bp的cfDNA片段的比例显着高于MR法(P = 0.0005),而与MPC提取的二核苷酸和三核苷酸结合的cfDNA比例显着低于(P = 0.0024和P = 0.0038)。
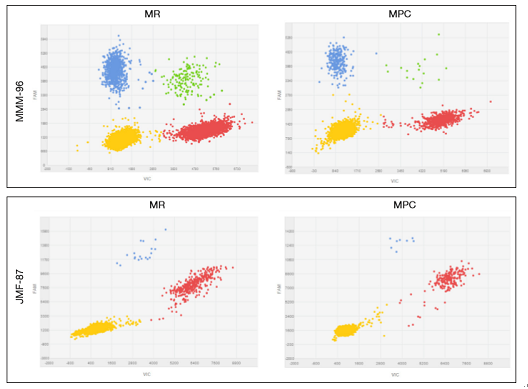
图2: 通过MPC和MR方法提取的配对cfDNA样品上肿瘤突变定量的代表性散点图。 (A)患者MMM-96,p.E746_A750delELREA突变用FAM(蓝色数据点)标记,而野生型cfDNA用VIC(红色数据点)标记; (B)患者JMF-87,p.T790M突变用FAM标记,野生型cfDNA用VIC标记。 cfDNA,循环游离DNA; MPC,MagNA Pure Compact; MR,Maxwell®RSC。
提取方法对肿瘤突变分析的影响
病理报告显示,在57例中,FFPE样本中有26例发生药物改变。其中17例对应于肺癌肿瘤中发现的EGFR突变,9例对应于结肠直肠肿瘤中发现的KRAS突变。在相应FFPE样品中发现可药用突变的26例患者中,买球网能够使用dPCR检测从19个血浆样品中分离的38个配对cfDNA样品中鉴定出的变化。在图2中显示了用于成对的cfDNA样品的肿瘤突变定量的代表性图。在剩余的在cfDNA上没有检测到肿瘤改变的情况下,在治疗期间提取血液样品,并且诊断患者具有部分应答或完全响应到RECIST v1.1标准。
在上述38个cfDNA样品中,30个对应于通过MR和MPC平行提取的15个血浆样品中配对的cfDNA样品。定义为突变体DNA拷贝相对于突变体和wt DNA拷贝的总和的比例的MAF及其所有分析样品的置信区间如表2所示。如图所示,平均来说,MAF在用MR和MPC,根据方法对cfDNA的肿瘤突变定量没有显着性差异(P = 0.8647),表明提取方法对cfDNA突变分析的影响较小。
最后,分析了MR和QCNA从四个血浆样品中分离出的8个cfDNA配对样品进行肿瘤突变检测和定量(表3)。如先前观察到的,配对cfDNA样品之间的MAF相似。
讨论
越来越多的证据支持在血液来源的样品如cfDNA(9-11)中可以有效检测肿瘤突变。此外,采集的相对容易程度和微创性质使得cfDNA成为纵向监测癌症疾病和对治疗反应的有吸引力的临床分析物(7,8)。因此,预期临床实验室将管理的“液体”样本数量将显着高于FFPE样本。在这种情况下,开发用于cfDNA隔离的自动化系统是一个重要的需要解决的问题。然而,提取阶段对于确保可靠的结果至关重要。值得注意的是,cfDNA具有一些特殊性,因为它们可以深刻地影响提取产率和下游应用。也就是说,cfDNA浓度通常很低,而且cfDNA高度片段化,其具有约180bp的短峰片段,其倍数似乎对应于核小体DNA(12,13)。
几项研究比较了不同的提取方法,用于分离血清/血浆样本中的cfDNA,确实证明提取方法可以显着影响cfDNA的产量(14-17)。类似地,买球网观察到MPC和MR提取方法在血浆样品中的cfDNA回收率显示出显着差异(P <0.0001)。在比较QCNA试剂盒和MR时,买球网发现后者在cfDNA产量方面并不优于前者,但由于它是一种自动化方法,它更简单和更快速。据买球网所知,上述方法比较在文献中以前没有报道过。此外,买球网的结果表明,测试的试剂盒显示单,二核小体DNA片段的恢复显着不同(表1)。应该在更大尺寸的队列中验证该观察结果,因为已经表明低分子量的cfDNA部分经常表现出指示肿瘤衍生的DNA的遗传畸变(17,18)。
如前所述(5),与非转移患者相比,买球网发现转移性疾病患者血浆中的cfDNA水平显着升高。此外,买球网已经观察到,在死亡患者中,cfDNA浓度随疾病进程特别高而增加(数据未显示)。平行地,已经描述了与健康个体相比,循环核小体在癌症患者中的浓度高得多(19-21)。有趣的是,据报道,在各种肿瘤类型中,肺癌与循环核小体的最高值相关(19,20)。类似地,买球网在大多数分析的样品中观察到寡核苷酸DNA梯形图(88%)。
核小体释放到循环中的机制被认为是由靶向治疗如酪氨酸激酶抑制剂诱导的凋亡性细胞死亡(22,23)。当追踪靶向治疗监测的cfDNA突变时,应考虑此信息。如果通过不同程序分离cfDNA可以影响更短和更长的cfDNA片段的恢复,则肿瘤突变定量可能受到提取方法的影响是合理的。然而,根据cfDNA提取方法,买球网没有发现MAF的显着差异。这可能部分是由于分析的样本数量较少,更大尺寸的队列将是特别有意义的,以澄清这个问题。值得注意的是,在一个样品(表2中的RTS-12)中,买球网无法检测到由MR分离的cfDNA中的药物改变,但在MPC提取的cfDNA中检测到。如上所述,已经表明,cfDNA的低分子量级分富集在肿瘤DNA中(18,19)。根据买球网的研究结果,虽然整个cfDNA提取产量显着较低(P <0.0001),MPC方法大小从150至200bp的cfDNA片段的回收率显着高于MR法(P = 0.0005)。这可以至少部分解释观察到的差异。
结论
在本文中买球网证明,根据评估的方法,cfDNA提取产率和高分子量和低分子量cfDNA级分回收率有显着差异。需要更大的研究来评估这些差异对下游应用的影响,例如用于靶向治疗监测的生物标志物测试或肿瘤突变跟踪。
致谢
作者希望感谢参与本研究的所有患者。
资助:本研究由西班牙科学与创新部卫生研究所卡洛斯三世和欧洲区域发展基金(拨款编号:PI13 / 01806和PIE14 / 00064)支持,罗梅罗由琼·罗德斯奖学金支持(授权号: JR14 / 00017)和CP博士前研究由JoseLuísCastaño基金会支持。
脚注
利益冲突:作者没有任何利益冲突声明。
道德声明:该研究由医院普埃尔塔耶罗伦理委员会批准(批准号:内部代码PI / 144-14)。病人获得书面知情同意书。
参考文献
1. International Agency for Research on Cancer, Cutado MP, Edwards B, et al. Cancer Incidence in Five Continents: Volume IX. Lyon: International Agency for Research on Cancer, 2008.
2. Siegel R, Ma J, Zou Z, et al. Cancer statistics, 2014. CA Cancer J Clin 2014;64:9-29.
3. American Society of Clinical Oncology. The state of cancer care in America, 2014: a report by the American Society of Clinical Oncology. J Oncol Pract 2014;10:119-42.
4. Swarup V, Rajeswari MR. Circulating (cell-free) nucleic acids--a promising, non-invasive tool for early detection of several human diseases. FEBS Lett 2007;581:795-9.
5. Leon SA, Shapiro B, Sklaroff DM, et al. Free DNA in the serum of cancer patients and the effect of therapy. Cancer Res 1977;37:646-50.
6. Aung KL, Board RE, Ellison G, et al. Current status and future potential of somatic mutation testing from circulating free DNA in patients with solid tumours. Hugo J 2010;4:11-21.
7. Crowley E, Di Nicolantonio F, Loupakis F, et al. Liquid biopsy: monitoring cancer-genetics in the blood. Nat Rev Clin Oncol 2013;10:472-84.
8. Dawson SJ, Tsui DW, Murtaza M, et al. Analysis of circulating tumor DNA to monitor metastatic breast cancer. N Engl J Med 2013;368:1199-209.
9. He C, Liu M, Zhou C, et al. Detection of epidermal growth factor receptor mutations in plasma by mutantenriched PCR assay for prediction of the response to gefitinib in patients with non-small-cell lung cancer. Int J Cancer 2009;125:2393-9.
10. Kuang Y, Rogers A, Yeap BY, et al. Noninvasive detection of EGFR T790M in gefitinib or erlotinib resistant nonsmall cell lung cancer. Clin Cancer Res 2009;15:2630-6.
11. Romero A, Acosta-Eyzaguirre D, Sanz J, et al.
Identification of E545k mutation in plasma from a PIK3CA wild-type metastatic breast cancer patient by array-based digital polymerase chain reaction: Circulatingfree DNA a powerful tool for biomarker testing in advance disease. Transl Res 2015;166:783-7.
12. Jahr S, Hentze H, Englisch S, et al. DNA fragments in the blood plasma of cancer patients: quantitations and evidence for their origin from apoptotic and necrotic cells. Cancer Res 2001;61:1659-65.
13. Suzuki N, Kamataki A, Yamaki J, et al. Characterization of circulating DNA in healthy human plasma. Clin Chim Acta 2008;387:55-8.
14. Repiská G, Sedláčková T, Szemes T, et al. Selection of the optimal manual method of cell free fetal DNA isolation from maternal plasma. Clin Chem Lab Med 2013;51:1185-9.
15. Fong SL, Zhang JT, Lim CK, et al. Comparison of 7 methods for extracting cell-free DNA from serum samples of colorectal cancer patients. Clin Chem 2009;55:587-9.
16. Holmberg RC, Gindlesperger A, Stokes T, et al. Akonni TruTip(®) and Qiagen(®) methods for extraction of fetal circulating DNA--evaluation by real-time and digital PCR. Kuhlmann JD, Schwarzenbach H, Wimberger P, et al. LOH at 6q and 10q in fractionated circulating DNA of ovarian cancer patients is predictive for tumor cell spread and overall survival. BMC Cancer 2012;12:325.
17. Schwarzenbach H, Eichelser C, Kropidlowski J, et al. Loss of heterozygosity at tumor suppressor genes detectable on fractionated circulating cell-free tumor DNA as indicator of breast cancer progression. Clin Cancer Res 2012;18:5719-30.
18. Holdenrieder S, Stieber P, Förg T, et al. Apoptosis in serum of patients with solid tumours. Anticancer Res 1999;19:2721-4.
19. Holdenrieder S, Stieber P, Bodenmüller H, et al. Nucleosomes in serum of patients with benign and malignant diseases. Int J Cancer 2001;95:114-20.
20. Holdenrieder S, Stieber P, Bodenmüller H, et al. Circulating nucleosomes in serum. Ann N Y Acad Sci 2001;945:93-102.
21. Costa DB, Halmos B, Kumar A, et al. BIM mediates EGFR tyrosine kinase inhibitor-induced apoptosis in lung cancers with oncogenic EGFR mutations. PLoS Med 2007;4:1669-79; discussion 1680.
22. Faber AC, Li D, Song Y, et al. Differential induction of apoptosis in HER2 and EGFR addicted cancers following PI3K inhibition. Proc Natl Acad Sci U S A 2009;106:19503-8.
版权声明:
本网站所有注明“来源:“买球APP”的文字、图片和音视频资料,版权均属于买球APP所有,非经授权,任何媒体、网站或个人不得转载,授权转载时须注明
“来源:买球APP”。本网所有转载文章系出于传递更多信息之目的,且明确注明来源和作者,不希望被转载的媒体或个人可与买球网联系,买球网将立即进行删除处理。
网友评论:
相关阅读
- > 《买球网拾萃》2023年12月刊:简述实验室信息管理系统发展历程
- > 《买球网拾萃》2023年10月刊:分析前标本处理对神经元特异性烯醇化酶(NSE)检测的影响
- > 《买球网拾萃》2023年8月刊:PSA 检测的标本处理、质量控制及其临床应用
- > 《买球网拾萃》2023年6月刊:真空采血管标本放置时间和温度对常见检验项目的影响
- > 《买球网拾萃》2023年4月刊:游离 DNA 管使用现状与展望
- > 《买球网拾萃》2023年2月刊:肝素管的临床适用检测项目与不适用项
- > 《买球网拾萃》2022年12月刊:血清分离技术及其对临床生化检验结果的影响
- > 《买球网拾萃》2022年10月刊:标本处理对心肌肌钙蛋白测定的影响
- > 《买球网拾萃》2022年8月刊:一种新型材料-水凝胶的合成及其应用
- > 《买球网拾萃》2022年6月刊:RFID 技术及其在医疗和标本管理中的应用与发展

